4期肠道菌群与代谢组学课题设计及研究策略研讨会2019(3月北京)

- 参会报名
- 会议
- 会议日程
- 会议嘉宾
- 参会指南
- 邀请函下载

|
4期肠道菌群与代谢组学课题设计及研究策略研讨会2019(3月北京)已过期会议时间: 08:00至 2019-03-24 18:00结束
|
 会议
会议

6.掌握肠道菌研究样本收集、保管、储存及预处理方法及关键注意事项;
9. 赠送份最近5年的高价值肠道菌群与代谢方面的国自然标书
北京班培训时间:2019/3/23-24 (22号报到)
北京班培训金泰绿洲大酒店,北京市东城区永外彭庄甲58号
学习班组织方
主办方:医药加科研培训中心
 会议日程
会议日程
(最终日程以会议现场为准)
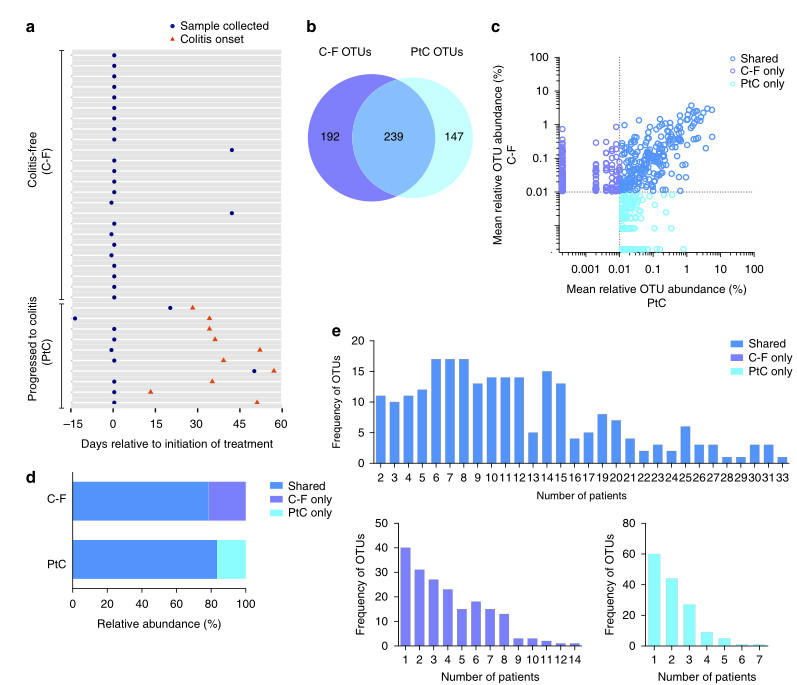
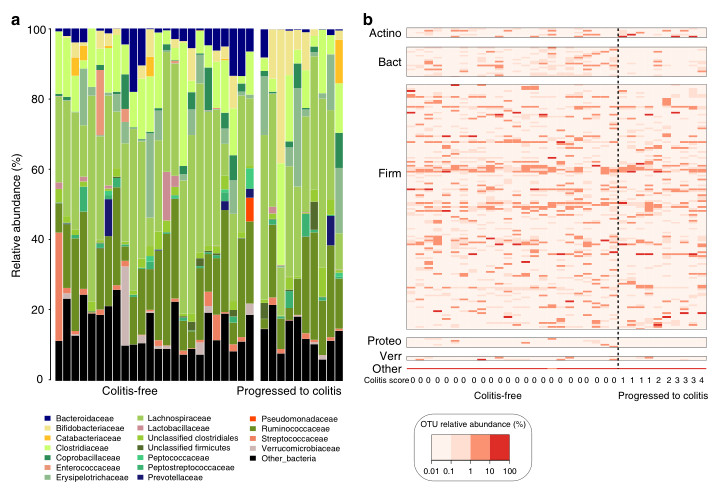
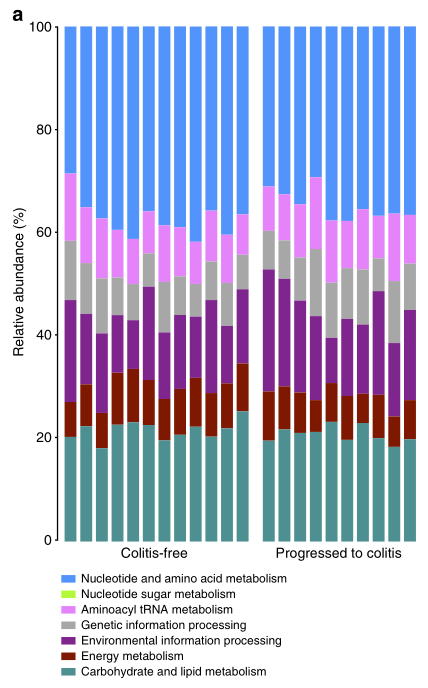
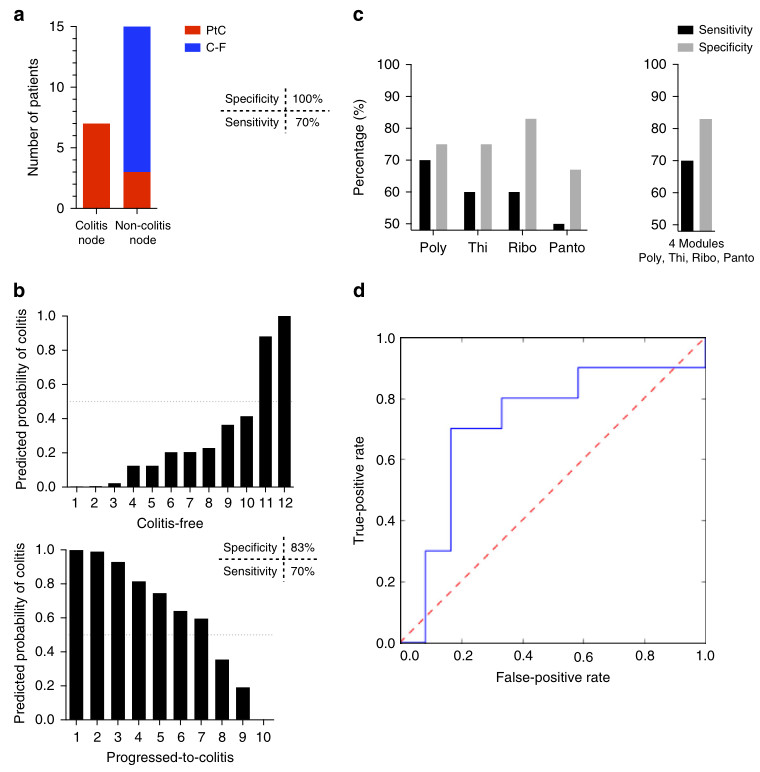
同群体、不同生长发育时期的肠道菌群; 研究某疾病发生发展进程中的群体的肠道菌群; 研究同群体多个身体部位的微生物; |
|||
功能宏基因组及应用; |
|||
 会议嘉宾
会议嘉宾
(最终出席嘉宾以会议现场为准)
专家:刘博士,长期专注于肠道微生物菌群变化与疾病相关性等方面研究,经验丰富、课程内容为原创设计,对此领域的科研思路以及最新研究动态熟悉掌握。从事相关科研课题设计工作多年,发表论文20余篇,其中10分以上2篇。多次应邀为大学、医院、科研单位进行相关主题讲座指导,具有丰富的课题设计和执行经验。并多次受邀在国际大会上发言,对微生物多样性、宏基因组、宏转录组、代谢组学等相关方向有深刻了解,且具有丰富讲课经验表达能力,通过两的课程定能让您对肠道微生物方面的课题设计及研究执行有很好的理解。
专家:吴教授,国内某高校教授,博士生导师。作为课题负责人先后主持肠道菌群与代谢组学研究国家级课题和部省级课题多项,并先后承担多项973计划课题中有关代谢组学方面的研究课题。所带领的团队主要采用代谢组学、生态学、生物信息学、分子生物学等多种宏观与微观相结合的方法,从宿主代谢与菌群微生态交互作用的角度对胃肠道疾病、神经退行性疾病的菌-肠-脑机制进行研究。目前已发表多篇SCI论文,在肠道菌群、代谢组学数据分析策略等相关领域受到了广泛关注和认可。
 参会指南
参会指南
会议门票场馆
学习费:3000元每位(学习费包含电子版教材、午餐,住宿费自理,保证学员完全掌握。)
4. 五人组团报名缴费,可额外带人免费注册!
5. 参加过医药加国自然撰写学习班的学员可优惠500元
 北京金泰绿洲大酒店
北京金泰绿洲大酒店
北京金泰绿洲大酒店地处南环陶然桥西南侧,毗邻陶然亭公园、安门、坛等旅游景点,紧邻北京南站,交通便利。
酒店餐厅不仅提供山西风味菜肴,还为您奉上川、鲁、粤等风味菜肴,让您齿颊留香、回味无穷。酒店拥有个可容纳150-220人的多功能厅及多种专业会议室,为您的会议和培训提供良好的办公环境。
温馨提示酒店与住宿:为防止极端情况下活动延期或取消,建议“异地客户”与manbext客户端下载客服确认参会信息后,再安排出行与住宿。退款规则:活动各项资源需提前采购,购票后不支持退款,可以换人参加。
 manbext客户端下载为本会议官方合作
报名平台,您可在线购票
manbext客户端下载为本会议官方合作
报名平台,您可在线购票
 会员折扣
该会议支持会员折扣 具体折扣标准请参见plus会员页面
会员折扣
该会议支持会员折扣 具体折扣标准请参见plus会员页面 会员返积分
每消费1元累积1个会员积分。 仅PC站支持。
会员返积分
每消费1元累积1个会员积分。 仅PC站支持。 会员积分抵现
根据会员等级的不同,每抵用1元可使用的积分也不样,具体可参见PLUS会员页面。 仅PC站支持。
会员积分抵现
根据会员等级的不同,每抵用1元可使用的积分也不样,具体可参见PLUS会员页面。 仅PC站支持。
.png)







